編輯 | 蘿蔔皮
生物技術行業一直在尋找完美的突變,將不同蛋白質的特性合成組合以達到預期的效果。可能有必要開發新的藥物或酶來延長酸奶的保質期,在野外分解塑膠,或使洗衣粉在低水溫下有效。
蛋白質序列的學習表示可以大大改進進行生物學預測的系統,並且還可能有助於揭示以前未發現的生物學資訊。如何選擇表示數據,對後續從中提取資訊的能力有根本性的影響。
機器學習有望從大型非結構化數據集(例如生物學中出現的數據集)中自動確定有效表示。然而,經驗證據表明,這些機器學習模型看似微小的變化會產生截然不同的數據表示,從而導致對數據的不同生物學解釋。這就引出了一個問題,即什麽才是最有意義的表示。
丹麥技術大學和哥本哈根大學的研究人員研究了這個問題,並探索了表示蛋白質序列的方法。他們主要探討了自然產生表征的兩個關鍵上下文(context):遷移學習和可解釋學習。
在遷移學習上下文中,研究人員證明了幾種當代實踐產生了次優的效能;在可解釋學習中,他們證明將表示幾何考慮在內可顯著提高可解釋性,並使用模型揭示了之前方法被掩蓋的生物資訊。
該研究以「 Learning meaningful representations of protein sequences 」為題,於 2022 年 4 月 8 日釋出在【 Nature Communications 】。

數據表示在生物數據的統計分析中起著至關重要的作用。從本質上講,表示是將原始數據提煉成一個抽象的、高級的、通常是低維的空間,該空間捕獲原始數據的基本特征。隨後可以將其用於數據探索,例如透過視覺化,或在可用數據有限的情況下進行特定任務的預測。
鑒於表示的重要性,說明生物學表示學習(表征)的興起並不奇怪,這是機器學習的一個子領域,其中表示與統計模型一起估計。特別是在蛋白質序列的分析中,過去幾年已經產生了許多研究,說明表征對於「如何從透過測序技術獲得的數百萬數據中自動提取關鍵資訊」十分重要。
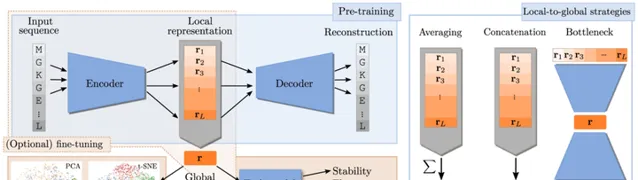
圖示:蛋白質序列的表示。(來源:論文)
雖然這些有希望的結果表明,表示學習可以對科學數據分析產生重大影響,但它們也帶來了一個問題: 什麽是好的表示?
表示學習的一個經典例子是主成分分析(PCA),它學習與原始數據線性相依的特征。當代技術消除了線性假設,轉而尋求高度非線性關系,通常透過使用神經網絡。
這在自然語言處理(NLP)中特別成功,其中單詞序列的表示是從大量線上文本資源中學習的,提取支持後續特定語言任務的語言的一般內容。這種詞序列模型的成功激發了其在生物序列建模中的套用,在遠端同源物檢測、功能分類和突變效應預測等套用領域取得了令人矚目的成果。
由於表征正在成為生物序列分析的重要組成部份,應該批判性地思考所構建的表征是否有效地捕獲了真正想要的資訊。研究人員討論了這個主題,重點是蛋白質序列,盡管許多見解也適用於其他生物序列。該工作由兩部份組成。
首先 ,考慮遷移學習設定中的表示;他們調查了網絡設計和訓練協定對結果表示的影響,並行現當前的幾種做法不是最理想的。
其次 ,研究了出於數據解釋目的而使用表示;結果表明,表示幾何的顯式建模使研究人員能夠提取穩健且可辨識的生物學結論。
該研究的結論闡明了,「什麽構成蛋白質的有意義表示」問題的部份答案。結論之一是問題本身沒有一個單一的通用答案,並且必須始終透過對表示目的的規範進行限定。
適合進行預測的表示,可能不是人類研究人員更好地理解潛在生物學的最佳選擇,反之亦然。因此,所有任務的單一蛋白質表示的誘人想法在實踐中似乎是行不通的。
設計有目的的表示
為給定任務設計表示需要反思研究者希望表示封裝哪些生物學特性。蛋白質的不同生物學方面將對表示提出不同的要求,但在表示中強制執行特定內容並不簡單。然而,研究人員可以透過
(1)選擇合適的模型架構;
(2)預處理數據;
(3)選擇合適的目標函數;
(4)將先驗分布放在模型的某些部份上來引導表示學習。
知情的網絡架構 可能難以構建,因為通常的神經網絡「構建塊」是相當基本的數學函數,不會立即與高級生物資訊相關聯。盡管如此,對長度不變序列表示的討論是一個簡單的例子,說明了人們如何為任務生物學的模型架構提供資訊。人們普遍認為,全域蛋白質特性與局部特性不是線性相依的。
因此,當允許模型學習這種非線性關系而不是依賴於局部表示的公共線性平均值時,模型效能顯著提高也就不足為奇了。將這個想法推廣到這裏探索的 Resnet 架構之外會很有趣,特別是結合最近的大規模基於轉換器的語言模型。
研究人員推測,雖然類似的「唾手可得的果實」可能仍然存在於當前套用的網絡架構中,但它們是有限的,需要更先進的工具將生物資訊編碼到網絡架構中。基於註意力的架構中的內部表示,已被證明可以恢復蛋白質之間已知的物理相互作用,從而為整合有關蛋白質中已知物理相互作用的先驗資訊開啟了大門。近期關於神經網絡中排列和旋轉不變性/等變性的研究工作均很有希望,盡管它們尚未在表示學習中進行詳盡的探索。
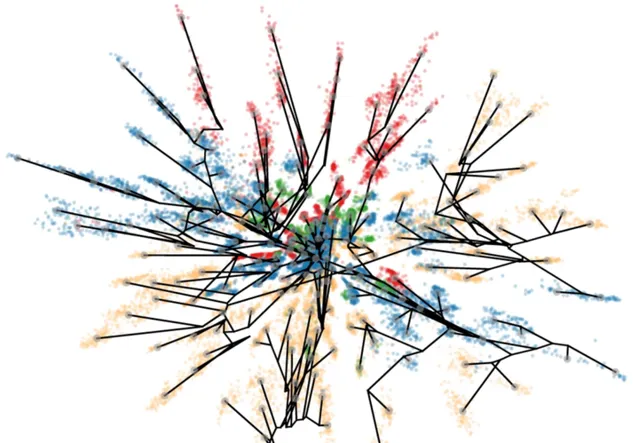
圖示:編碼到潛在表示空間中的系統發育樹。(來源:論文)
數據預處理和特征工程 在當代「端到端」表示學習中不受歡迎,但它仍然是模型設計的重要組成部份。特別是,使用來自計算生物學的大量現有工具進行預處理,是將現有生物學知識編碼到表示中的一種有價值的方法。
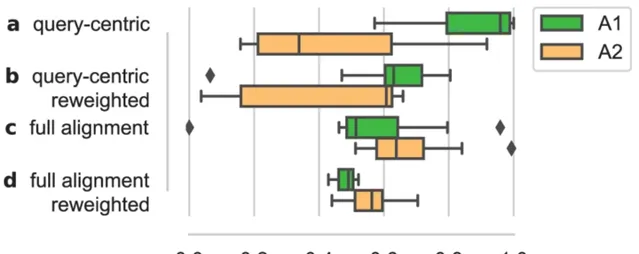
圖示:對齊預處理對表示可靠地解碼回蛋白質序列的能力的影響。(來源:論文)
在對對齊的蛋白質序列進行訓練時,可以看到無監督模型的表示能力有了顯著提高,因為這註入了關於一組序列中可比較序列位置的先驗知識。雖然近期的研究工作越來越多地致力於直接從數據中學習此類訊號的技術,但仍不清楚多重對齊所提供的優勢是否可以被這些方法完全代替。
其他預處理技術,例如序列的重新加權,目前也依賴於對齊序列。這些例子表明,如果學界向「端到端」學習邁進得太快,可能會因為放棄現有工具中多年的經驗,而出現將嬰兒和洗澡水一起扔出去的荒誕事件。
相關的目標函數 對於任何學習任務都至關重要。盡管表示學習通常使用重建損失進行,但研究人員證明根據此目標的最佳表示對於任何特定的遷移學習任務通常都不是最佳的。這表明應該根據下遊任務特定的效能來選擇表示的超參數,而不是在保留集上的重建效能。
然而,這是一個微妙的過程,因為在下遊任務上最佳化表示模型的參數與過度擬合的高風險有關。研究人員預計,將大型無監督數據集上的重建目標與半監督學習環境中的特定任務目標相結合的原則性技術,將在該領域提供實質性的好處。
資訊先驗 可以施加比硬架構約束編碼的偏好更軟的偏好。VAE 中的高斯先驗就是這樣一個例子,盡管它的偏好不受生物資訊的引導,這似乎是一個錯失的機會。在對 β-內醯胺酶的研究中,研究人員觀察到一種類似於蛋白質家族前進演化所跨越的系統發育樹的表示結構。最近旨在強調數據階層的雙曲線先驗可能有助於更清楚地提出這種前進演化結構。當賦予合適的黎曼度量時,潛在表示可以更好地反映生物學,因此使用相應的幾何先驗可能很有價值。

圖示:β-內醯胺酶蛋白質家族的潛在嵌入,由分類學在門級進行顏色編碼。(來源:論文)

圖示:VAE 中 β-內醯胺酶表示之間的最短路徑(測地線)。(來源:論文)
適當地分析表示
即使盡了最大的努力將先驗知識納入表征,仍然必須非常小心地解釋它們。研究人員強調表示空間中距離的特定範例,並強調看似自然的歐幾列特距離具有誤導性。現代機器學習方法中編碼器和解碼器的非線性,意味著表示空間通常是非歐幾裏得的。
該團隊已經證明,透過將觀察空間的預期距離以黎曼度量的形式帶入表示空間,他們獲得的測地線距離與系統發育距離的相關性明顯優於透過通常的歐幾裏得檢視可以獲得的距離。這是一個令人興奮的結果,因為黎曼檢視帶有一組類似於加法和減法的自然算子,因此表示可以在操作上進行。研究人員希望這對蛋白質工程等有價值,因為它提供了一種結合不同蛋白質表達的操作方法。
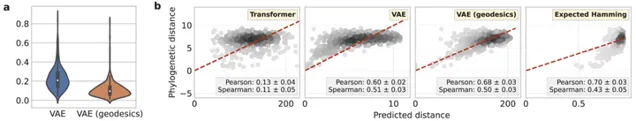
圖示:測地線在潛在空間中提供更穩健和更有意義的距離。(來源:論文)

圖示:兩個蛋白質序列之間的插值。(來源:論文)
在這項研究中,研究人員僅對變分自編碼器的潛在空間進行幾何分析,由於其從固定維度潛在空間到固定維度輸出空間的平滑對映,因此非常適合。由於他們無法從順序語言模型中的聚合全域表示進行解碼,這一事實阻礙了超出單個蛋白質家族的擴充套件。
一個自然的問題是,他們提出的瓶頸策略是否可以使這種分析成為可能。如果是這樣,它將為定義潛在空間中遠端同源物之間有意義的距離提供新的可能性,並可能允許改進蛋白質之間的 GO/EC 註釋轉移。
最後,幾何分析帶來了一些與蛋白質無關的含義。它表明,在歐幾裏得螢幕上將潛在表示繪制為點的常用視覺化可能具有高度誤導性。因此,科學家認為需要能夠忠實反映表示幾何的視覺化技術。分析還表明,下遊預測任務可能會從利用幾何結構中獲益,盡管標準神經網絡架構尚不具備這種能力。
論文連結: https://www. nature.com/articles/s41 467-022-29443-w
相關報道: https:// phys.org/news/2022-05-m achine-potentials-proteins.html











