「我們對大部份RNA的結構幾乎一無所知。」
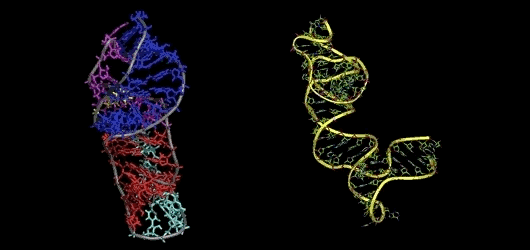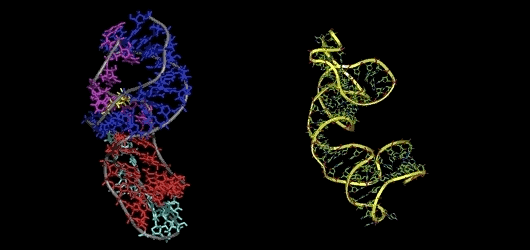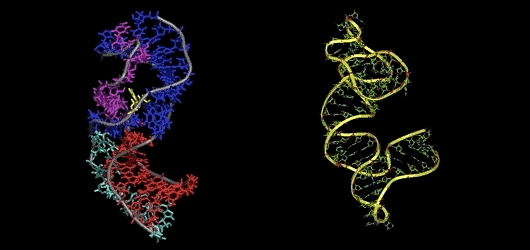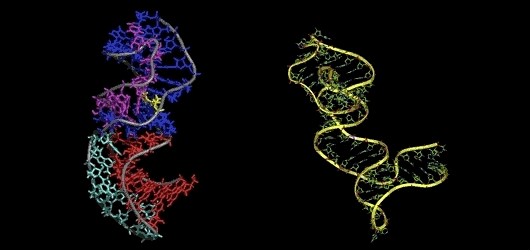
半個世紀以來,確定生物分子的三維結構一直困惑著科學家,也是生物學的重大挑戰之一。難就難在,RNA折疊成復雜三維結構的形狀很難透過實驗或計算來確定。

而現在,美國史丹福大學透過新型AI演算法準確預測出RNA三維結構,可謂意義重大!今天,Science 封面刊登了這項研究的最新成果,由史丹福大學在讀博士生 Stephan Eismann 和 Raphael Townshend,以及電腦副教授 Ron Dror共同完成。他們利用目前先進的神經網絡技術,成功開發出了一種全新 RNA 三維結構預測模型——ARES。

從原子入手,機器學習演算法預測RNA結構!
RNA 分子和蛋白質一樣,會折疊成三維 (3D) 結構,來執行一系列功能,如催化反應、基因表現、調節先天免疫和感知小分子。RNA的三維結構十分重要,有助於理解RNA發揮作用的機制、設計合成 RNA 和發現 RNA 靶向藥物。

人類基因組轉錄成 RNA 的部份是蛋白質編碼的 30 倍左右,然而,我們對RNA結構的認知還遠遠不夠,已知的RNA結構只是蛋白質結構的不到1%。本文的創新之處在於引入了一種新的機器學習方法,開發出一個人工智能模型ARES(Atomic Rotationally Equivariant Scorer)來預測RNA結構,準確率極高!
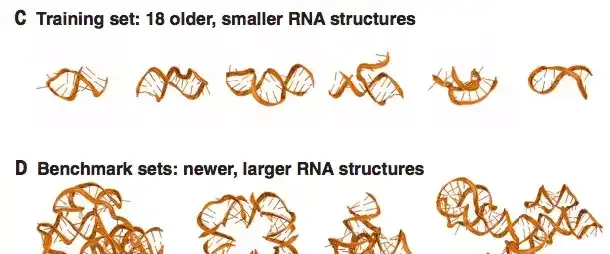
而且,研究人員僅用了 18 個已知的 RNA 結構進行訓練。
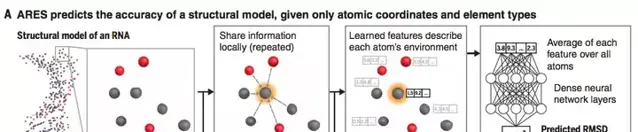
更厲害的是,ARES無需對RNA的顯著特征進行假設,也就是說,它沒有關於雙螺旋、堿基對、核苷酸或氫鍵的先入為主的概念。這就使ARES可以適用於任何類別的分子系統!
ARES包括一組已知 RNA 結構的基序和這些結構的替代(錯誤)變體。透過調整參數,ARES可以了解每個原子的功能和幾何排列,和不同原子間的相對位置。這也是與其他模型不同的一點。然後,神經網絡各層可以計算不同粗細尺度的特征,來辨識堿基對、RNA螺旋的最佳幾何形狀、三維空間結構。
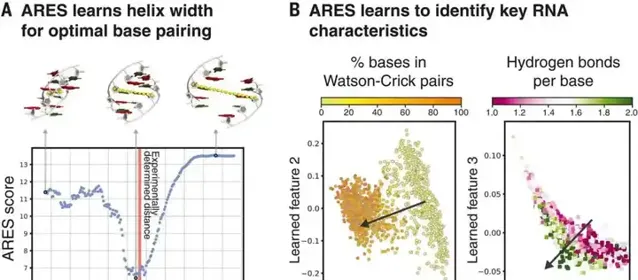
具體來看,ARES網絡的初始層旨在辨識結構基序(生物大分子中的保守序列),也就是說,基序的特點是在訓練過程中學習的,而不是預先設定的。然後,根據前一層的特征和周圍原子的幾何排列,計算出每一個原子的特征。第一層的唯一輸入是每個原子的 3D 座標和化學元素種類。
這些初始網絡層的架構認識到,給定的結構基序彼此間通常有不同的定位,而且,較粗尺度的基序(如螺旋)通常包括更細尺度的基序(如堿基對)的特定排列。另外,每一層都是旋轉和平移等變的,也就是說,輸入的旋轉或平移在輸出時有相應的變換。以上特點可確保將已辨識基序的方向和位置傳遞到ARES網絡的下一層,下一層則使用此資訊來辨識較粗尺度的基序。ARES 還可以預測全域內容,同時詳細捕獲局部結構基序和原子間相互作用。因為它的初始層在本地收集資訊,而其余層則匯總所有原子的資訊。測試得出,ARES可以準確辨識結構模型。
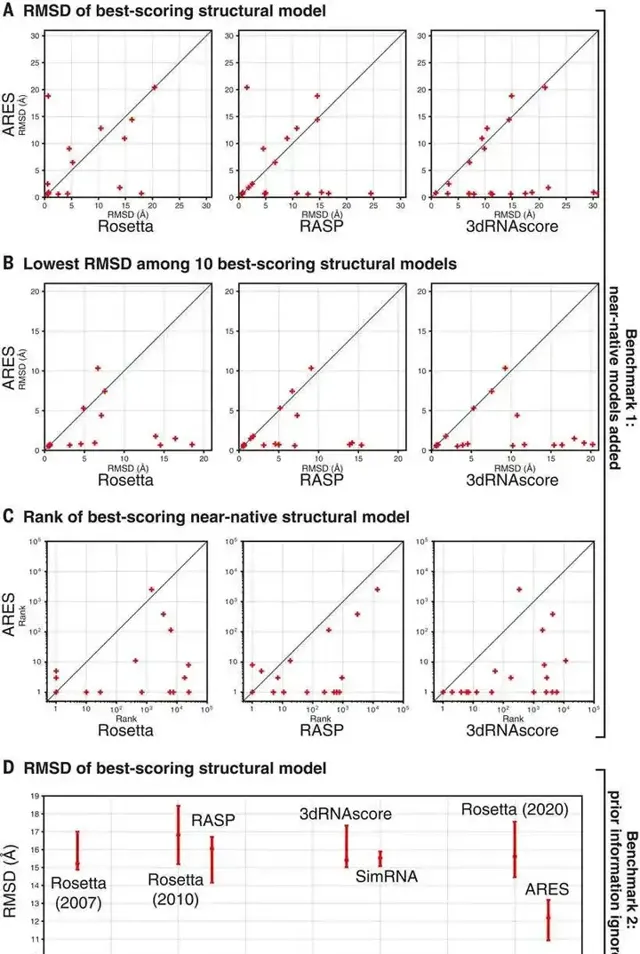
對RNA結構做出準確預測。
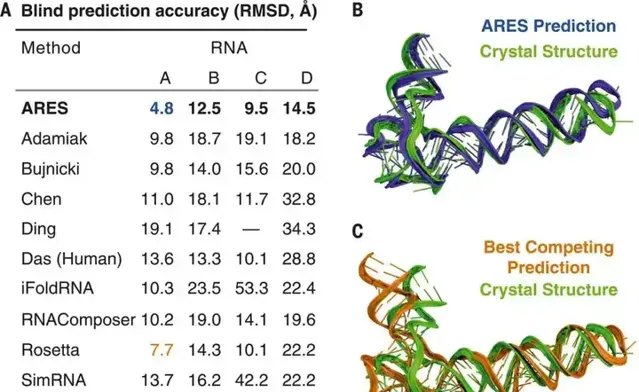
作者介紹
文章一作是Raphaël Townshend,1993年生於加拿大蒙特利爾,是史丹福大學電腦科學博士,目前創辦了一家設計新分子和藥物的人工智能公司,並擔任CEO。

個人主頁:https:// raphael.tc.com/2010-201 4 年,他曾在加州大學柏克萊分校獲得電子工程與電腦科學學士學位。Townshend對機器學習、結構生物學、高效能計算、電腦視覺這些領域的研究感興趣。他曾做過表情辨識的一個CV專案。將呈現在受試者臉部的灰度影像中的6種表情分類 (喜悅、悲傷、厭惡、憤怒、驚訝、恐懼)

他利用 Gabor 濾波器對輸入影像進行一對多線性支持向量機訓練。並使用了現有的標準數據集,比如,Cohn-Kanade 和 JAFFE,以及自己收集的47個主題的數據集。
參考資料:
http:// science.sciencemag.org/ content/373/6558/1047
http:// science.sciencemag.org/ content/373/6558/964
https:// news.stanford.edu/2021/ 08/26/ai-algorithm-solves-structural-biology-challenges/
https:// twitter.com/raphaeljlt/ with_replies










